Seu teclado pode estar cheios de bactérias. Veja pesquisa
Professora do Revisamed divulga estudo sobre contaminação em computadores
Artigo publicado na Revista Médica de Minas Gerais pela doutora em Ciências da Saúde, área de concentração Saúde Brasileira, Patrícia Guedes Garcia, professora da equipe do Curso Preparatório para Residência Médica, Revisamed, destaca a contaminação microbiana em computadores utilizados em um hospital público da cidade de Juiz de Fora, Minas Gerais.
Teclados de computadores e mouses fabricados de materiais plástico favorecem a permanência de bactérias patogênicas. No estudo foi possível observar altas taxas de contaminação por bactérias patogênicas nos teclados e mouses do hospital, apresentando grande relevância o controle destes microrganismos nas IRAS e na disseminação de bactérias patogênicas, sendo um desafio e uma prioridade para as instituições de saúde.
O artigo conclui que as infecções relacionados à Assistência à Saúde representam um problema de saúde pública, sendo causadas por diversos microorganismos. Representam um importante problema de saúde pública, tanto nos países desenvolvidos como em países em desenvolvimento, pois aumentam as taxas de morbidade e de mortalidade, prolongam o período de internação e elevam os custos hospitalares.
Leia abaixo artigo na íntegra
Avaliação da contaminação microbiana em computadores utilizados em um hospital público da cidade de Juiz de Fora
Assessment of microbial contamination in computers used in a public hospital in the city of Juiz de Fora
Patrícia Guedes Garcia1, Gabriela Souza Barbosa2, Leandro Oliveira de Morais3, Laura Alcântara Damianse4, Leonardo Romaniello Gama de-Oliveira5
RESUMO
Introdução
Infecções Relacionadas à Assistência à Saúde representam um problema de saúde pública, sendo causadas por diversos microrganismos. Teclados de computadores são fabricados com materiais plásticos que favorecem a permanência de bactérias patogênicas.
Objetivos
Pesquisar e avaliar o perfil de resistência de bactérias patogênicas isoladas em teclados e mouses utilizados em um hospital de Juiz de Fora. Métodos: Foram coletadas 110 amostras (55 de teclados e 55 de mouses), com swab estéril em meio Stuart. Estas foram inoculadas em caldo BHI, incubadas por 24 horas em estufa de aerobiose, à 35ºC. Foram repicadas em Ágar Manitol Salgado, Ágar MacConkey e Ágar Sangue e incubadas por 24-48 horas, em aerobiose, a 35º ± 2ºC. Colônias presentes nos meios de cultura foram identificadas por provas bioquímicas e fisiológicas e submetidas ao teste de sensibilidade aos antimicrobianos.
Resultados
Foram analisados 55 teclados e 55 mouses utilizados nos setores do hospital. Das 110 amostras coletadas, 39 (35,45%) apresentaram crescimento bacteriano, sendo 21 (19,09%) em teclados e 18 (16,36%) em mouses. Com relação aos microrganismos encontrados, houve prevalência de: Enterobacter cloaceae, S. aureus, Stenotrophomonas maltophilia, Acinetobacter sp, Enterococcus sp., Escherichia coli, Klebsiella pneumoniae, e Klebsiella oxytoca.
Foram analisados 55 teclados e 55 mouses utilizados nos setores do hospital. Das 110 amostras coletadas, 39 (35,45%) apresentaram crescimento bacteriano, sendo 21 (19,09%) em teclados e 18 (16,36%) em mouses. Com relação aos microrganismos encontrados, houve prevalência de: Enterobacter cloaceae, S. aureus, Stenotrophomonas maltophilia, Acinetobacter sp, Enterococcus sp., Escherichia coli, Klebsiella pneumoniae, e Klebsiella oxytoca.
Conclusões
Foi possível observar altas taxas de contaminação por bactérias patogênicas nos teclados e mouses do hospital, apresentando grande relevância o controle destes microrganismos nas IRAS e na disseminação de bactérias patogênicas, sendo um desafio e uma prioridade para as instituições de saúde.
Palavras-chave: Computadores. Infecção Hospitalar. Resistência Microbiana a Medicamentos.
Introdução
Infecções Relacionadas à Assistência à Saúde (IRAS) referem-se àquelas adquiridas durante a prestação dos cuidados de saúde, após admissão do paciente, durante a internação ou após a alta.1 Representam um importante problema de saúde pública, tanto nos países desenvolvidos como em países em desenvolvimento, pois aumentam as taxas de morbidade e de mortalidade, prolongam o período de internação e elevam os custos hospitalares.1,2
Apesar dos importantes avanços alcançados no controle das infecções, estudos demonstram a emergência do problema globalmente, pelo aumento acentuado da frequência e da gravidade dos casos de IRAS, o que pode ter sido favorecido pela disseminação de microrganismos resistentes, pelo fato da não adesão dos profissionais às medidas de biossegurança, a sobrecarga de trabalho e, por vezes, o número reduzido de recursos humanos.3,4
O que são as IRAS
As IRAS são causadas por diversos microrganismos, com predomínio de bactérias Gram-negativas. Aquelas de maior relevância clínica e epidemiológica em ambientes hospitalares são as enterobactérias como Escherichia coli, Proteus sp., Serratia marcescens, Enterobacter sp. e Klebsiella pneumoniae e os microrganismos não fermentadores de glicose como Pseudomonas sp., Stenotrophomonas sp., Burkholderia sp. e Acinetobacter sp.5
A contaminação do ambiente hospitalar por bactérias Gram-positivas como Staphylococcus aureus resistente a meticilina (MRSA) e Enterococcus resistente a vancomicina (VRE), especialmente em situação epidêmica, tem sido frequentemente descrita.
A proporção de superfícies hospitalares contaminadas com MRSA tem variado consideravelmente de 1% a 27% em quartos hospitalares a 64% de superfícies em Unidades de Queimados com pacientes portando MRSA. Por conseguinte, a contaminação ambiental pode contribuir na transmissão de microrganismos epidemiologicamente importantes na gênese de infecções hospitalares.6
Superfície ambientais contaminadas
Há evidências na literatura de que superfícies ambientais contaminadas com microrganismos podem contribuir para a transmissão desses patógenos quando associados aos cuidados de saúde.6 Essas superfícies desempenham um papel significante na ocorrência da transmissão cruzada, pois atuam como fontes constantes de contaminação, inclusive pelo contato das mãos dos profissionais com os pacientes e pelo contato direto do paciente com material ou ambiente contaminado.6,7
O avanço técnico-científico permitiu um aumento dos sistemas de informatização clínica, tendo por objetivo a automação de todas as rotinas administrativas do hospital.8,9 Alguns estudos têm enfatizado a contaminação de objetos inanimados em uso nos ambientes hospitalares, assim como telefones, computadores, estetoscópios e inclusive aqueles equipamentos presentes nos leitos dos pacientes.8
Métodos
Avaliou-se a presença de microrganismos em teclados e mouses de computadores utilizados em um hospital público de Juiz de Fora, Minas Gerais.
As amostras foram coletadas entre fevereiro e março de 2015, com swab estéril com meio Stuart (Labor® Swab). Este foi friccionado contra a superfície dos teclados. Após a coleta, as amostras foram encaminhadas ao laboratório de microbiologia da Faculdade de Ciências Médicas e da Saúde de Juiz de Fora.
As amostras foram inoculadas em caldo BHI (Mbiolog diagnósticos Ltda. ®), incubadas por 24 horas em estufa de aerobiose, (Fanem® 502 São Paulo – Brasil), a 35ºC ± 1. Após este período, foram repicadas em Ágar Manitol Salgado (RenyLab®, Química e Farmacêutica), Ágar Sangue 5% (RenyLab®, Química e Farmacêutica) e Ágar MacConkey (RenyLab®, Química e Farmacêutica), incubadas por 24-48 horas, em aerobiose, a 35º ± 1ºC e realizadas as provas bioquímicas para a identificação de cada espécie.10,11
Para identificação de Staphylococcus aureus foi utilizada a fermentação do manitol, catalase e coagulase. Já para Enterococcus sp, utilizaram-se as provas da catalase, NaCl 6,5% e bile esculina.10,11
Para identificar os bastonetes Gram-negativos foram utilizados prova do OF, IAL, citrato, arginina, ornitina, lisina, motilidade, oxidase e polimixina B.10,11
As bactérias identificadas foram submetidas ao teste de sensibilidade aos antimicrobianos (TSA) pelo método de Ágar Difusão em Disco, de acordo com a padronização do CLSI 2014 – Clinical Laboratory Standards Institute.12
Os resultados apresentados consideraram somente o isolamento de S. aureus, bastonetes Gram-negativos fermentadores e não fermentadores e Enterococcus sp.
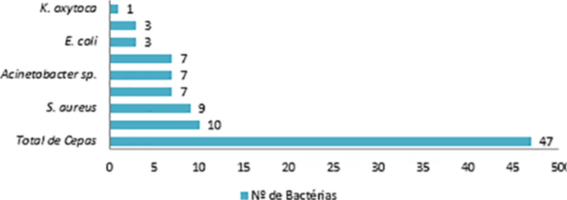
Com relação aos microrganismos bacterianos encontrados, dentre as 39 amostras positivas de teclados e mouses, 47 cepas foram identificadas, com prevalência de 10 cepas (21,3%) de Enterobacter cloacea, 9 (19,1%) de S. aureus, 7 (14,9%) de Stenotrophomonas maltophilia, 7 (14,9%) de Acinetobacter sp., 7 (14,9%) de Enterococcus sp., 3 (6,4%) de Escherichia coli, 3 (6,4%) de Klebsiella pneumoniae, e 1 (2,1%) de Klebsiella oxytoca como demonstrado na Figura 2.
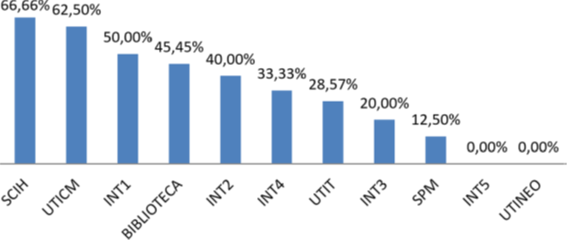
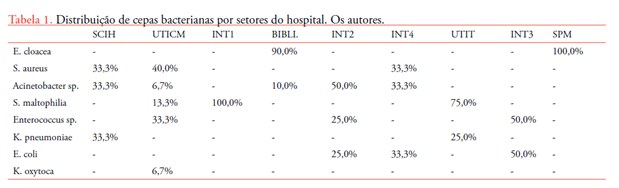
Foram encontrados na SCIH 33,33% de Acinetobacter sp., 33,3% de K. pneumoniae e 33,3% de S. aureus. Na UTICM a prevalência foi de 40,0% de S. aureus, 33,3% de Enterococcus sp., 13,3% S. maltophilia, 6,7% de K.oxytoca e 6,7% de Acinetobacter sp.. Na INT1 o crescimento foi de 100,0% de S. maltophilia, e na biblioteca foi de 90,0% de E. cloacea e 10,0% de Acinetobacter sp.. Na INT2 foi de 50% de Acinetobacter sp., 25% de Enterococcus sp.e 25% de E. Coli, e na INT4 de 33,3% S. aureus, 33,3% Acinetobacter sp. e 33,3% E. Coli. Na UTIT 75,0% foi S. maltophilia e 25,0% de K. pneumoniae. Na internação 3, 50,0% de E. coli e 50,0% de Enterococcus sp., e na SPM 100,0% de E. cloacea, conforme Tabela 1.
Foi realizado o Teste de Sensibilidade aos Antimicrobianos (TSA), para avaliar o perfil de resistência das bactérias encontradas. Os antimicrobianos testados foram de acordo com a padronização do CLSI 2014, conforme Tabela 2.
Para família Enterobacteriaceae, testaram-se os antimicrobianos: piperacilina + tazobactam, ceftazidma, ceftriaxona, cefepime, aztreonam, ertapenem, meropenem, imipenem, gentamicina, amicacina, sulfametoxazol + trimetoprin e levofloxacin. As cepas de K. pneumoniae foram, uma betalactamase de espectro estendido (ESBL) e duas Klebsiella pneumoniae carbapenemase (KPC), e apresentaram 33,40% de resistência para piperacilina+tazobactam e gentamicina, 66,60% de resistência para meropenem, imipenem, ertapenem e sulfametoxazol + trimetoprim e 100% de resistência para ceftazidima, ceftrixona, cefepime e aztreonam. A cepa
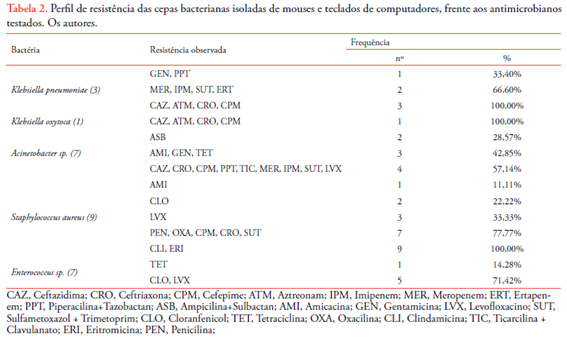
de K. oxytoca foi ESBL, apresentando 100% de resistência para ceftazidima, ceftrixona, cefepime e aztreonam. As cepas de E. cloacae e E. coli, não apresentaram resistência a nenhum antimicrobiano testado.
Para Acinetobacter sp foram testados: piperacilina + tazobactam, ceftazidma, ceftriaxona, cefepime, aztreonam, meropenem, imipenem, gentamicina, amicacina, sulfametoxazol + trimetoprin, levofloxacin, ampicilina + sulbactam, ticarcilina + clavulanato, tetraciclina. O resultado para as cepas isoladas foi de 28,57% de resistência a ampicilina + sulbactam, 42,85% para amicacina, gentamicina e tetraciclina e 57,14% para ceftazidima, ceftriaxona, cefepime, piperacilina + tazobactam, ticarcilina + clavulanato, meropenem, imipenem, sulfametoxazol + trimetoprim e levofloxacin.
Todas cepas de S. maltophilia foram sensíveis a levofloxacin e sulfametoxazol + trimetoprin.
Para S. aureus, foram testadas penicilina, oxacilina, ceftriaxona, cefepime, cloranfenicol, levofloxacin, amicacina, clindamicina, eritromicina, sulfametoxazol + trimetoprin, teicoplamina e linezolida, e para Enterococcus sp., tetraciclina, cloranfenicol, ampicilina, penicilina, levofloxacin, linezolida, teicoplamina e vancomicina. O perfil de resistência foi 11,11% para amicacina, 22,22% para cloranfenicol, 33,33% para levofloxacin, 77,77% para penicilina, oxacilina, cefepime, ceftriaxona e sulfametoxazol + trimetoprim, e 100% para clindamicina e eritromicina. Dentre as nove cepas de S. aureus, 77,77% (7) apresentaram resistência a cefoxitina, MRSA.
Para as cepas de Enterococcus sp. foram testadas ampicilina, penicilina, linezolida, teicoplamina, vancomicina, tetraciclina, levofloxacin e cloranfenicol. O perfil de resistência foi de 14,28% para tetraciclina e 71,42% para levofloxacin e cloranfenicol.
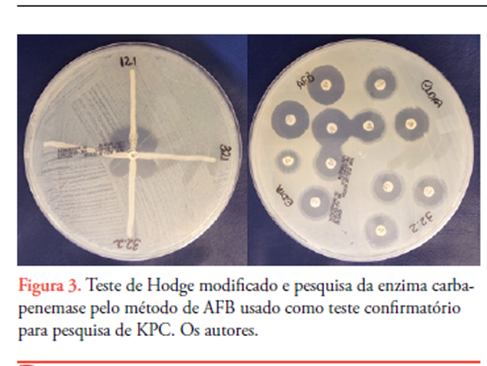
Para as duas cepas de K. pneumoniae resistentes aos carbapenêmicos, foram feitos testes fenotípicos de Hodge e ácido fenilborônico (AFB), para detecção da enzima Klebsiella pneumoniae carbapenemase (KPC), segundo o CLSI 2014 e a nota técnica da ANVISA 001/2013.13,14 Tanto o teste de Hodge, quanto a pesquisa de carbapenemases pelo método do ácido fenilborônico foram positivos para presença de KPC (Figura 3).
DISCUSSÃO
A contaminação por microrganismos pode ocorrer na maior parte dos ambientes, sendo esta intimamente ligada à higiene local. Sabe-se que a sobrevivência de tais patógenos no ambiente pode variar de minutos a meses, sendo que objetos de uso rotineiro, tais como os teclados, podem se tornar focos de transmissão a hospedeiros.14
Em todas as amostras foram observados crescimentos de Staphylococcus sp coagulase negativa (SCN). Os SCN são facilmente encontrados, visto que faz parte da microbiota da pele e não representa, necessariamente, risco à saúde dos usuários hígidos.15,16,17
Detectou-se, através do TSA, a presença de MRSA em 7 (77,77%) cepas de S. aureus isoladas. Um estudo microbiológico de 2011 realizado em superfícies de leitos da UTI de um hospital escola em Três Coroas – MG, totalizou 63 amostras e das 48 amostras positivas para S. aureus, 29 (60,4%) foram resistentes à meticilina.
A resistência do S. aureus aos antimicrobianos tem sido desenvolvida por mutações em seus genes ou pela aquisição de genes de resistência de outras bactérias da mesma espécie (ou ate de outras). A literatura descreve que a resistência a meticilina é determinada por um gene cromossômico (mecA) bacteriano, o qual codifica modificações no receptor do betalactâmico, estimulando a produção de uma proteína ligadora de penicilina (PPB2a) com baixa afinidade pelo antimicrobiano, resultando na resistência.
Geralmente, a resistência que ocorre por mutação gera uma alteração no sitio de ação do antimicrobiano, enquanto a resistência por aquisição de genes de resistência frequentemente envolve a inativação ou a destruição da droga, sendo transmitida por plasmídeos e transposons.18
Das sete cepas de Enterococcus sp. encontradas, todas apresentaram sensibilidade à vancomicina. Um estudo de 2006 analisou 50 amostras de teclados em um hospital e isolou 12 cepas de Enterococcus sp., nenhuma apresentou resistência à vancomicina.9 Outro estudo, de 2013, concebido em um leito de uma UTI adulta em um hospital do Vale do Rio Pardo – RS, encontrou-se apenas uma cepa de Enterococcus sp., sendo esta sensível à vancomicina, o que corrobora com os resultados evidenciados.19
Enterococcus sp. pode sobreviver por longos períodos em diversas superfícies e solos, dentro de esgotos e hospitais, crescendo a temperaturas entre 10 e 45°C de ambientes básicos ou ácidos, isotônicos ou hipertônicos, manifestando alta resistência às drogas (vancomicina, penicilina, gentamicina, tetraciclina, eritromicina e teicoplanina), devido não só a genes cromossômicos que codificam resistência, mas também a plasmídeos e transposons conjugativos.8
A colonização ou infecção por VRE tem sido associada a uma variedade de fatores, incluindo tempo de internação hospitalar, doença de base e transplante hepático. Pacientes colonizados por VRE carregam o microrganismo em sua microbiota intestinal e podem permanecer colonizados por até dois anos. Após sua introdução em determinado hospital, o Enterococcus sp. apresenta grande capacidade de disseminação, criando um perfil de endemicidade que dificulta tentativas de erradicação posterior.19
Observou-se o crescimento de cepas da família Enterobacteriaceae, sendo E. cloacae o mais isolado, com prevalência de nove cepas (21,3%), que não apresentaram resistência aos antimicrobianos testados. Infecções causadas pelos microrganismos do gênero Enterobacter são raras nos pacientes imunocompetentes.
Nesse trabalho foram isoladas três cepas de E. coli, que não apresentaram resistência em nenhum dos antimicrobianos testados. Das três amostras de K. pneumoniae isoladas, uma foi ESBL e duas foram KPC. A resistência aos carbapenêmicos tem sido codificada por diversos genes, principalmente blaKPC.14
A produção de ESBL é um mecanismo de resistência em enterobactérias. O tratamento de infecções causadas por cepas produtoras de ESBL oferece um substancial desafio à terapia antimicrobiana, pois as ESBLs hidrolisam penicilinas, cefalosporinas de todas as gerações e monobactâmicos, minimizando opções terapêuticas. Ademais, o uso contínuo e inadequado de agentes antimicrobianos pode induzir à seleção de cepas multirresistentes.20
Detectou-se também uma cepa de K. oxytoca produtora de ESBL, que apresentou 100% de resistência as cefalosporinas de 3ª e 4ª geração e aztreonam. Um estudo realizado de 2012 pesquisou ESBL em E. coli e Klebsiella sp isoladas em superfícies inanimadas em um hospital do Peru. Das 125 amostras obtidas, isolou-se 20 cepas de E.coli, 19 cepas de K. pneumoniae e 4 cepas de K. oxytoca. Cinco cepas de E. coli e 11 cepas de Klebsiella sp foram produtores de ESBL, com exceção de uma cepa de E. coli e uma K. oxytoca, todas as outras cepas foram resistentes às cefalosporinas de 3ª geração, aztreonam e cefepime.21
As duas cepas K. pneumoniae isoladas resistentes aos carbapenêmicos foram submetidas a pesquisa fenotípica de KPC através dos testes de Hodge modificado e AFB, sendo que os dois testes foram positivos para a presença de KPC.
A enzima KPC é produzida por bactérias Gram-negativas (Enterobactérias), e sua detecção em isolado bacteriano confere resistência aos antimicrobianos carbapenêmicos, além de inativar penicilinas, cefalosporinas e monobactâmicos. É importante salientar que os carbapenêmicos compreendem uma classe amplamente utilizada no tratamento de infecções envolvendo Enterobacteriaceae multirresistente. Vários são os mecanismos de resistência que podem impedir a ação dos carbapenêmicos, e a resistência surge, ocasionalmente, da combinação de impermeabilidade da membrana com betalactamases cromossômicas (AmpC) ou de amplo espectro (ESBL), gerando pequena hidrolise de carbapenêmicos e, ainda, alteração nos canais de porina, que modificam a ação e a penetração dos fármacos.22,23,24
Estudos têm comprovado a emergência dos bastonetes gram-negativos não fermentadores como causadores de IRAS mundialmente, sendo Pseudomonas sp. e Acinetobacter sp. os mais frequentes. O crescimento microbiano predominante de Acinetobacter sp. multirresistente nas superfícies de materiais e equipamentos mostra a relevância deste patógeno no contexto hospitalar, uma vez que as estas cepas tem capacidade de sobreviver em superfícies secas por períodos longos de tempo, mantendo sua capacidade de multiplicação e infectividade.
Neste estudo foram encontradas sete cepas (14,9%) de Acinetobacter sp., que apresentaram elevado perfil de resistência para todas as cefalosporinas testadas e aos carbapenêmicos. Um estudo realizado no hospital de emergência no Sul do Brasil, em 2010 pesquisou a presença dos genes responsáveis pelo mecanismo de resistência e verificou-se que as cepas de Acinetobacter sp. encontradas apresentavam sensibilidade reduzida as cefalosporinas de terceira geração (ceftazidima e ceftriaxona), além de apresentarem resistência ao imipenem.25
Foram encontradas sete cepas (14,9%) de S. maltophilia, sendo que todas apresentaram sensibilidade a levofloxacin e a sulfametoxazol + trimetoprim. Em 2011, um estudo avaliou o perfil de sensibilidade das cepas de S. maltophilia isoladas no Hospital das Clínicas da Faculdade de Medicina da Universidade de São Paulo, e, o perfil de sensibilidade apresentado pelos isolados de S. maltophilia neste estudo foi compatível com o característico desse agente, apresentando baixos níveis de sensibilidade aos antimicrobianos betalactâmicos e betalactâmicos associados a inibidores de betalactamase e melhor sensibilidade a sulfametoxazol + trimetoprim e fluoroquinolonas.26
Os resultados encontrados no presente estudo são semelhantes àqueles encontrados pela maioria dos autores que estudaram e isolaram bactérias patogênicas das superfícies inanimadas dos mais variados ambientes, e se tratando de superfícies secas, revela uma maior persistência no plástico, material usado na fabricação de teclados e mouses. Amostras microbiológicas de superfícies podem ser úteis nas investigações epidemiológicas que sugerem o ambiente ou superfícies como sendo possíveis reservatórios ou fontes de transmissão de doenças nosocomiais.
Conclusão
Foi possível observar contaminação por bactérias patogênicas nos teclados e mouses nos diversos setores do hospital. A contaminação de computadores corrobora a hipótese de que superfícies muito tocadas se tornam mais contaminadas. Tal premissa reforça a ideia de que muitas vezes os profissionais, após tocar um paciente, não se atêm à importância da higienização das mãos e retomam as atividades sem se dar conta da possibilidade de disseminar microrganismos.
As cepas bacterianas isoladas apresentaram alta taxa de resistência bacteriana, sendo possível observar mecanismos de resistência como ESBL, KPC e MRSA.
Dessa forma, considerando todas as possibilidades dos microrganismos isolados neste trabalho de manifestarem resistência aos antimicrobianos, favorecendo a infecção em pacientes imunocomprometidos, é de suma importância que os componentes da comissão de infecção hospitalar incluam entre os materiais vistoriados em suas inspeções também os teclados e mouses. Isso deve ser associado à orientação dos trabalhadores das unidades de saúde sobre quais protocolos seguir para evitar futuros casos de infecções nosocomiais.
Referências
1. Oliveira AC, Silva MDM, Garbaccio JL. Vestuário de profissionais de saúde como potenciais reservatórios de microrganismos: Uma revisão integrativa. Texto Contexto Enferm. 2012; 21 (3): 684-91.
2. Albuquerquer AM, Souza APM, Torquato IMB, Trigueiro JVS, Ferreira JA, Ramalho MAN. Infecção Cruzada no Centro de Terapia Intensiva á Luz da Literatura. Rev Ciênc Saúde Nova Esperança. 2013; 11 (1):78-87.
3. Oliveira AC, Damasceno QS. O papel do ambiente hospitalar na disseminação de bactérias resistentes. Rev Epidemiol Control Infect. 2012; 2 (1):28-31.
4. Correal JCD, Marques EA, Guilherme WL, Leão RS, Damas-co PV. Infecções por Staphylococcus aureus: mudança do perfil epidemiólogico no Hospital Universitário Pedro Ernesto. Rev HUPE. 2013; 12 (3):31-46.
5. Coelho Neto GT, Moraes FC, Monteiro Neto V, Figuereirêdo PMS. Detecção de enterobactérias em superfícies de uma unidade mista de saúde no município de São Luís, Maranhão, Brasil. Rev Invest Biomed Uniceuma. 2010; (2): 77-84.
6. Ferreira AM, Andrade D, Almeida MTG, Cunha KC, Rigotti MA.Colchões do tipo caixa de ovo: um reservatório de Staphylococcusaureus resistente à meticilina?. Rev Esc Enferm USP. 2011; 45(1):161-6.
7. Fernando FSL, Ferreira AM, Colombo TE, Rubio FG, Almei-da MTG. Contaminação por fungos antes e após limpeza e desinfecção do colchões hospitalares. Acta Paul Enferm. 2013; 26(5):485-91.
8. Rodrigues AG, Viveiros MAWB, Barroso IMO, Cavalcante AP, López AMQ. Contaminação bacteriana em teclados de computadores utilizados em hospital universitário do nordeste do Brasil. Medicina (Ribeirão Preto). 2012; 45 (1): 39-48.
9. Binatti VB, Costa CRM, Tórtora JCO. Patógenos hospitalares resistentes em teclados de computadores. J Bras Med. 2006; 90: 28-37.
10. Koneman EW. Diagnóstico Microbiológico: texto e atlas. 6th ed. Rio de Janeiro: Guanabara Koogan; 2008.
11. Oplustil CP, Zoccoli CM, Tobouti NR. Procedimentos Básicos em Microbiologia Clínica. 3 rd ed. Rio de Janeiro: Sarvier; 2010.
12. Clinical Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility test for bacteria that grow aerobically 2014.
13. Agência Nacional de Vigilância Sanitária (ANVISA). Nota Técnica n° 1/2013 – Medidas de prevenção e controle de infecções por enterobactérias multiresistentes. Brasília (DF): ANVISA; 2013.
14. Alves JLB, Costa RM, Braoios A. Teclados de computadores como reservatórios de microrganismos patogênicos. J Health Sci Inst. 2014;32(1):7-11.
15. Moraes CL, Ribeiro NFG, Costa DM, Furlan VG, Palos MAP, Vasconcelos LSNOL. Contaminação de equipamentos e superfícies de unidades de terapia intensiva de uma maternidade pública por Staphylococcus Coagulase Negativa. Rev Patol Trop.
2013; 42 (4): 387-94.
16. Sousa Junior FC, Nunes EWF, Nascimento ED, Oliveira SM, Melo MCN, Fernandes MJBC. Prevalência de Staphylococcus spp resistentes à meticilina isolados em uma maternidade escola da cidade de Natal, Estado do Rio Grande do Norte. Rev Soc Bras Medicina Tropical 2009; 42 (2):179-82.
17. Pereira PMA, Castro EAR, Pereira JAA, Tórtora JCO. Resistência aos antimicrobianos em estafilococos coagulase-negativa isolados em hemocultura. Jornal Brasileiro de Medicina. 2007; 93 (5/6): 13-6.
18. Santos AL, Santos DO, Freitas CC, Ferreira BLA, Afonso IF, Rodrigues CR et al. Staphylococcus aureus: visitando uma cepa de importância hospitalar. J Bras Patol Med Lab. 2007; 43 (6): 413-23.
19. Renner JDP, Carvalho ED. Microrganismos isolados de superfícies da UTI adulta em um hospital do Vale do Rio Pardo – RS. Rev Epidemiol Control Infect. 2013; 3(2):40-4.
20. Lago A, Fuentefria SR, Fuentefria DB. Enterobactérias produtoras de ESBL em Passo Fundo, Estado do Rio Grande do Sul, Brasil. Rev Soc Bras Med Trop. 2010; 43 (4): 430-4.
Rivera-Jacinto MA. Betalactamasas de espectro extendido emcepas de Escherichia coli y Klebsiella sp.aisladas de reservorios
21. inanimados en umhospital del norte del Perú. Rev Esp Quimioter. 2012; 25(2):161-3.
22. Seibert G, Hörner R, Meneghetti BH, Righi RA, Dal Forno NL, Salla A. Infecções hospitalares por enterobactérias produtoras de Klebsiella pneumoniae carbapenemase em um hospital escola. Einstein. 2014; 12(3):282-6.
23. Soares VM. Emergência de Klebsiella pneumoniae produtora de carbapenemase (KPC) em um hospital terciário. J Bras Patol Med Lab. 2012; 48(4): 251-3.
24. Dienstmann R, Picoli SU, Meyer G, Schenkel T, Steyer J. Avaliação fenotípica da enzima Klebsiella pneumoniae carbapenemase (KPC) em Enterobacteriaceae de ambiente hospitalar. J Bras Patol Med Lab. 2010; 46 (1): 23-7.
25. Laranjeira VDS, Marchetti DP, Steyer JR, Corção G, Picoli SU. Pesquisa de Acinetobacter ap. e Pseudomonas aeroginosa produtores de metalo-β-lactamase em hospital de emergência de Porto Alegre, estado do Rio Grande do Sul, Brasil. Rev Soc Bras Med Trop. 2010: 43(4); 462-4.
26. Rodrigues LS, Di Gioia TSR, Rossi F. Stenotrophomonas maltophilia: resistência emergente ao SMX-TMP em isolados brasileiros. Uma realidade?. J Bras Patol Med Lab. 2011; 47(5):
511-7.